分子植物科学卓越创新中心李轩研究组完成涵盖五大植物类群的150 物种参考代谢组及其数据库
文章来源:分子植物科学卓越创新中心 | 发布时间:2023-11-14 | 【】 【】
2023年11月11日,中国科学院分子植物科学卓越创新中心李轩团队在国际学术期刊nucleic acids research在线发表了题为“refmetaplant: reference metabolome database for plants across five major phyla”的研究成果。这项研究阐述了植物“参考代谢组”概念,发展了参考代谢组图谱的组装方法,完成构建了五大植物类群: 苔癬植物、石松植物、蕨类植物、裸子植物和被子植物等类群的150多个植物物种的参考代谢组图谱。同时还开发了一系列与基因组分析工具(如blast)相似的代谢组分析工具(如refmetablast),为研究植物代谢组多样性和代谢物功能,提供了一个强大的数据共享和分析平台。
植物“参考代谢组”是指一个植物物种所有代谢物的总和。一个植物物种“参考代谢组”与代谢物之间的关系,类似于植物物种“参考基因组”与基因之间的关系。植物在亿万年进化过程中,发展了惊人的化学多样性和代谢复杂性。植物对复杂多变的环境适应, 形成种类繁多、结构高度复杂的代谢产物,这些代谢产物在植物生长、发育、生理调节和环境适应方面起着至关重要的作用。据估计,绿色植物的代谢产物数量,包括初级代谢物和次级代谢物达到百万以上。针对植物代谢组分析、解析代谢产物功能的挑战,近年来随着代谢组检测技术的发展,如高分辨率质谱与超高效液相色谱相结合的分析技术,能够以前所未有的高灵敏度、高分辨率、高准确性和高通量来检测代谢物,使在全组学规模的植物代谢物检测成为可能。与30年前多生物基因组计划开始时缺乏“参考基因组”和分析工具的状况类似,目前植物代谢组学研究的关键瓶颈是缺乏高质量的植物“参考代谢组”及其相关分析工具。
针对植物代谢组学研究这些关键问题,研究团队通过采集五大植物类群的多物种/多器官组织样本,基于高性能液质代谢组实验检测技术和开发的人工智能深度代谢组分析算法,首次完成构建涵盖绿色植物五大类群(苔藓植物、石松植物、蕨类植物、裸子植物和被子植物)的150 植物物种的参考代谢组及其数据库:refmetaplant。研究的主要成果包括:1)涵盖植物五大类群150 植物物种的参考代谢组;每个物种的参考代谢组 (.msr 或 .mgf 文件格式) 可在平台分析或下载。2)对每个植物物种的参考代谢组,提供基于人工智能分析的代谢物注释。3)用于代谢物注释的标准化合物谱图库及开发的一系列用于植物代谢组分析的工具,例如“lc-ms/ms query”“refmetablast”和“compoundlibblast”等。
refmetaplant旨在提供标准化的植物参考代谢组,为植物代谢及植物生理相关研究提供关键支持。作为植物代谢组的综合数据库平台,refmetaplant将成为植物代谢组学研究的核心资源,同时还将促进代谢组学领域的数据交流共享与合作。refmetaplant可以在https://www.biosino.org/refmetadb 免费使用。
分子植物卓越中心时晗、吴雪婷、朱艳、江涛为论文共同第一作者,李轩研究员、陈萍博士为共同通讯作者。感谢上海辰山植物园王正伟老师、江苏里下河地区农业科学研究所李爱宏研究员、肖宁研究员和刘建菊博士、上海植物园张映雪老师、河南农业大学陈锋教授、中国科学院东北地理与农业生态研究所冯献忠研究员和高金珊博士、分子植物卓越中心张鹏研究员和巫永睿研究员提供或者帮助采集植物物种材料。中国科学院营养与健康研究所张国庆研究员,分子植物卓越中心公共技术服务中心徐晓燕、胡文利和荆莲艳老师提供了大量j9九游会登录入口首页旧版的技术支持。该研究工作获得了中国科学院先导计划和国家自然科学基金的支持。
论文链接:
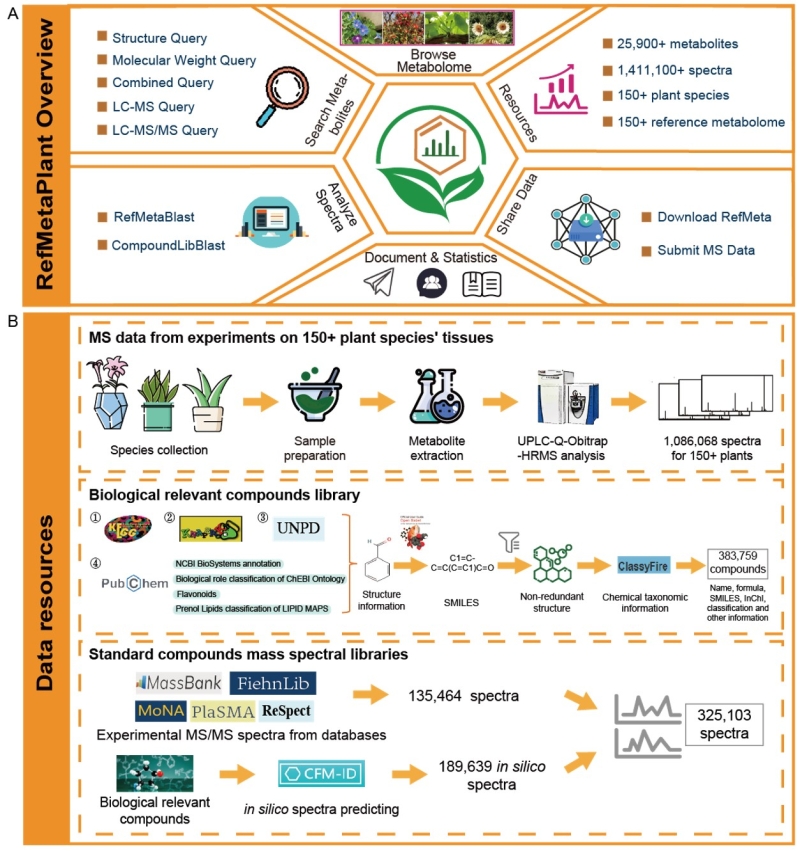
图1:refmetaplant数据库资源和工具介绍
(a) refmetaplant功能概述。(b) refmetaplant的数据资源及其工作流。
图2:植物物种:callistemon rigidus参考代谢组的图形解释
