数据驱动的水库抗生素抗性基因科学发现
文章来源:城市环境研究所 | 发布时间:2023-10-30 | 【】 【】
抗生素抗性基因(antibiotic resistance genes, args)在环境中的产生、扩散和传播,是“同一健康(one health)”框架体系下城市环境研究的重点领域之一。水作为城市人类活动与流域自然过程相互作用的关键环境介质,是多维空间尺度下args在“人类—动物—环境”之间扩散和传播的纽带。随着人类在城市聚集和活动强度增加,流域人工和自然水生态系统受args影响的过程更加复杂。尽管流域水生态系统有关的args研究随之迅速增加,但从“同一健康”框架体系来看,仍需“全数据(one data)”视角下多维尺度的探索。
水库由人类建设并使之惠益于人类,是一类用于“人—水”关系调节的人工水生态系统,被视为流域尺度上维系“同一健康”的关键基础设施。在“同一健康”框架体系下,水库被认为是args在城市水系统产生、扩散和传播的重要“调节器”。简言之,水库通过供水和灌溉这两个基本途径调节城市水系统args的传播与健康风险。因此,阐释水库args的形成机制与风险模式,对加强水库生态系统本身的管理以及降低args的健康风险均有至关重要的科学意义。
数据驱动发现(data-driven discoveries)是科学认知过程在研究数据不断累积的驱动下形成新发现的一种整合研究范式。例如,单个水库或局地若干水库args的研究结论难以直接外推到分布于全球各地的数以万座水库。因而,随着对args研究的水库数量不断增加,水库args数据整合将驱动该主题科学认知的反复验证和逐渐深化。对此,中国科学院城市环境研究所联合牛津大学、新墨西哥州立大学等单位研究人员,根据水库args研究的数据结构,改进了合作团队之前构建的“提取-分组-统计”等模块组成的数据挖掘方法,形成了适用于水库args科学发现的数据驱动路径(图1)。这一数据驱动路径将数据视为“果子”,并将路径本身以“果树”形状进行形象化表示。该路径改进的模块和相应科学发现以data-driven discoveries on widespread contamination of freshwater reservoirs by dominant antibiotic resistance genes为题发表在《water research》。
水库args数据驱动路径整合了60个水库中290个样本所检出的args组成丰度数据,覆盖了来自亚洲、欧洲、中东和南美洲共4个地理区域的40个城市。整合的数据集包含args的9个主要类型,包括氨基糖苷类、 -内酰胺类、氯霉素类、大环内酯-林可酰胺-链霉菌素b(mlsb)、磺胺类、四环素类、万古霉素类、多重耐药以及其它类型抗性基因。该数据集由210个水样本和80个沉积物样本构成,并以水库和样点为基本单元在数据矩阵中分别进一步关联了水库基本特征和理化参数等信息。该数据驱动路径的构建,为较大尺度阐释水库args污染格局、形成机制及风险模式提供了方法和数据支撑。
在污染格局方面,args已对水库造成了广泛的污染,但其类型和丰度在各大洲以及各水库之间各不相同。不同水库检出args的数量差异跨越1~2个数量级并且其类型各有差异。亚洲地区的水库水体受args污染程度普遍较高,并且其丰度较欧洲等地区高出一个数量级。水库水体和沉积物受args污染的调控因素明显不同,其调控因素之间的交互作用存在线性或非线性增强的统计学规律。在风险模式方面,富含有机质的小型水库水体在寒冷季节易遭受抗性基因污染,而较大库容和低水位水库沉积物在干旱春季的污染程度更严重(图2)。
尽管数据驱动路径所支撑的60个水库args污染和风险研究与单个或局地若干水库研究相比有了较大尺度的科学发现,但并不意味着上述结论已具有普适性。在“同一健康”框架体系和“全数据”视角下,面对全球数以万座计的水库,已有水库args数据驱动的科学发现依然是“冰山一角”。随着未来水库args研究的持续增加,数据类型和结构将分别更加丰富和复杂,涉及的时空尺度会更长、更广。因此,已构建的数据驱动路径也将难以直接应用,未来对数据分组模块和统计模块方法体系的更新应该进一步考虑到数据持续增加带来的尺度依赖性和因果涌现性。
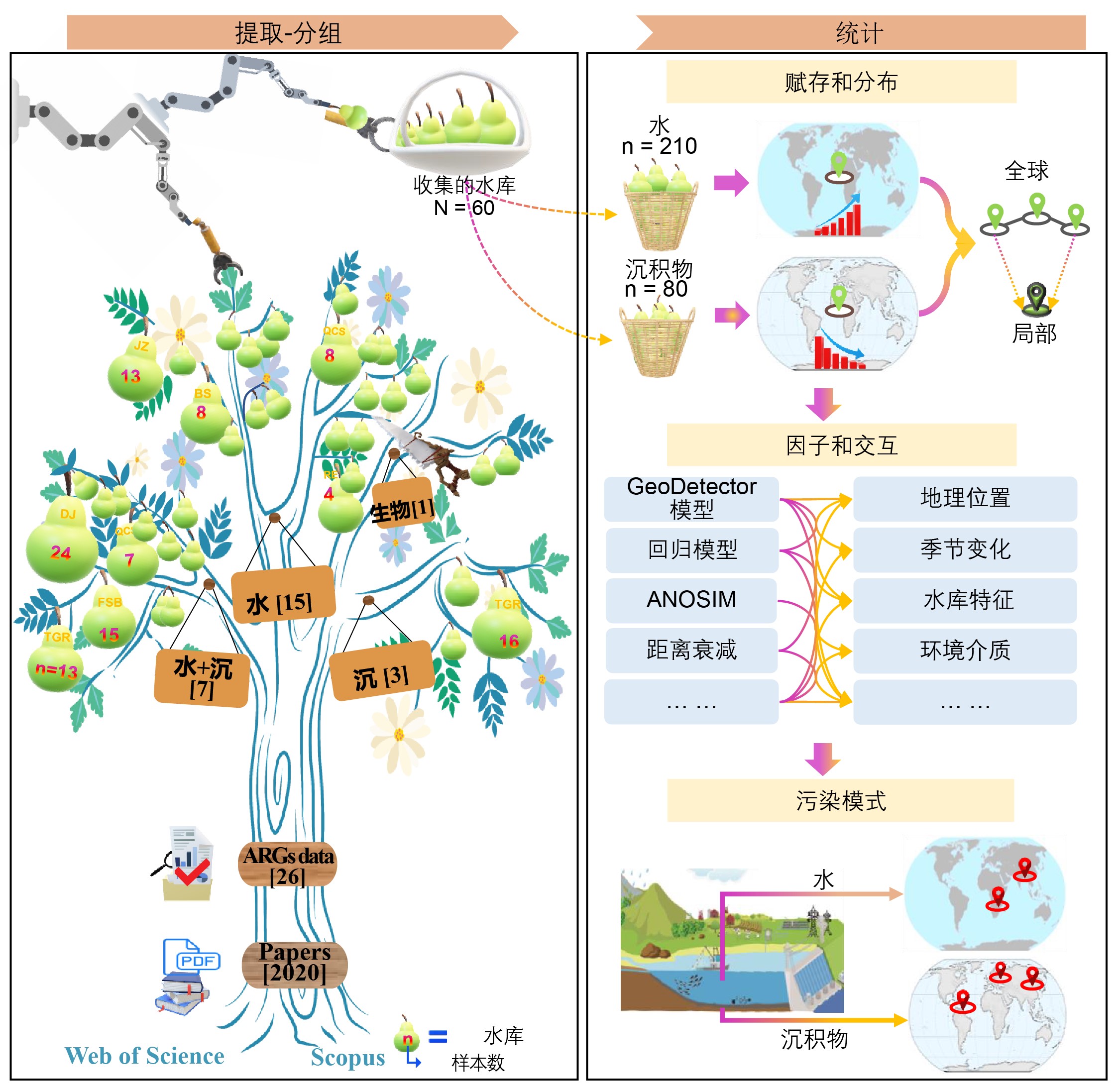
图1 面向水库args科学发现的数据驱动路径
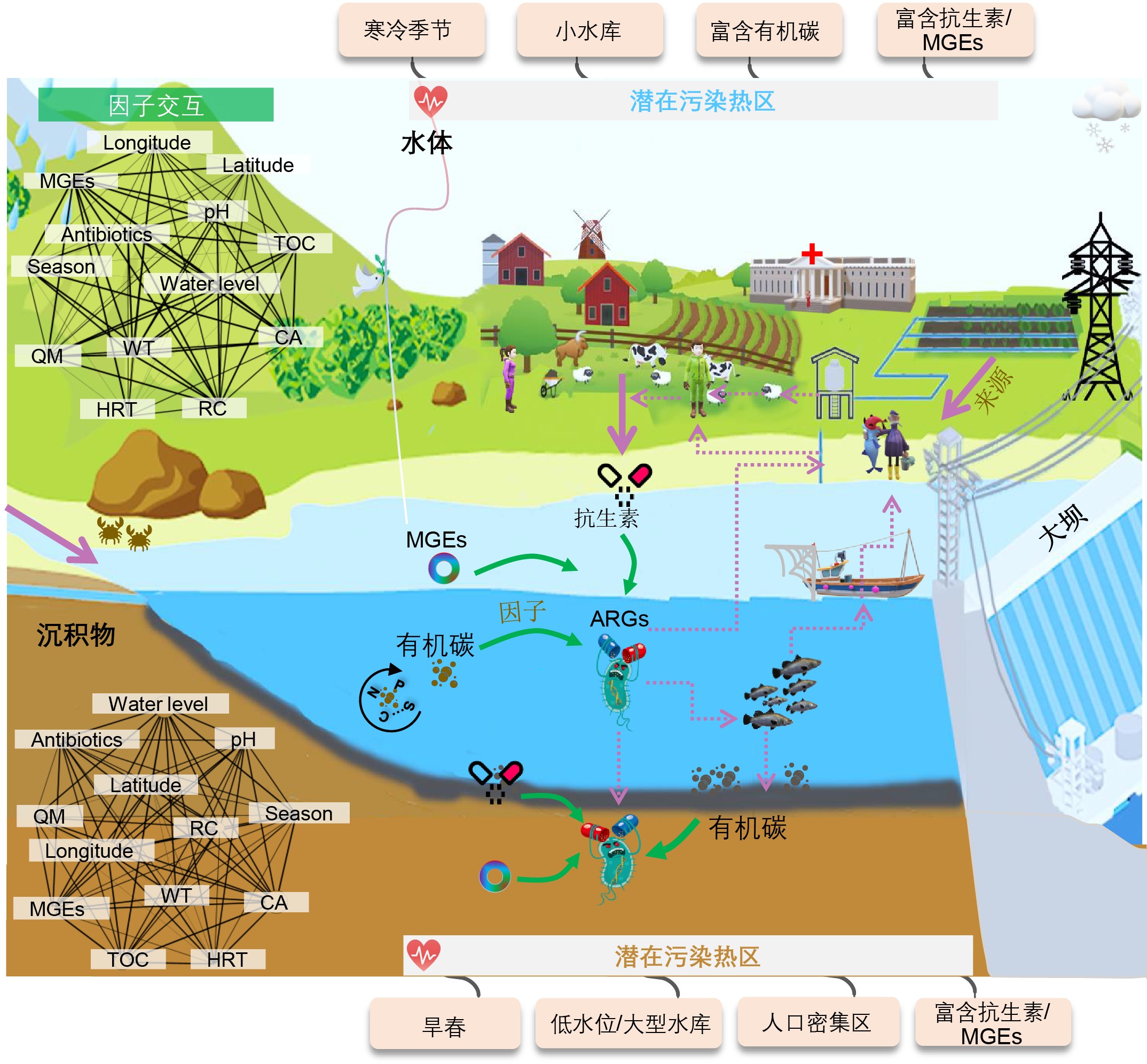
图2 水库args形成机制与风险模式
相关资料:
1. guo, z.f., boeing, w.j., xu, y.y., borgomeo, e., liu, d., zhu, y.g. (2023) data-driven discoveries on widespread contamination of freshwater reservoirs by dominant antibiotic resistance genes. water res. 229, 119466.
2. guo, z.f., boeing, w.j., xu, y.y., borgomeo, e., mason, s.a. and zhu, y.g. (2021) global meta-analysis of microplastic contamination in reservoirs with a novel framework. water res. 207, 117828.
3. zhu, y.g., gillings, m., simonet, p., stekel, d., banwart, s., penuelas, j. (2018) human dissemination of genes and microorganisms in earth's critical zone. glob. chang. biol. 24(4), 1488-1499.
4. zhu, y.g., zhao, y., li, b., huang, c.l., zhang, s.y., yu, s., chen, y.s., zhang, t., gillings, m.r., su, j.q. (2017) continental-scale pollution of estuaries with antibiotic resistance genes. nat. microbiol. 2, 16270.
5. mart nez, j.l. (2008) antibiotics and antibiotic resistance genes in natural environments. science 321(5887), 365-367.
6. berendonk, t.u., manaia, c.m., merlin, c., fatta-kassinos, d., cytryn, e., walsh, f., et al. (2015) tackling antibiotic resistance: the environmental framework. nat. rev. microbiol. 13(5), 310-317.
7. martinez, j.l., coque, t.m., baquero, f. (2015) what is a resistance gene? ranking risk in resistomes. nat. rev. microbiol. 13(2), 116-123.
8. hernando-amado, s., coque, t.m., baquero, f., martinez, j.l. (2019) defining and combating antibiotic resistance from one health and global health perspectives. nat. microbiol. 4(9), 1432-1442.
9. larsson, d.g.j., flach, c.f. (2022) antibiotic resistance in the environment. nat. rev. microbiol. 20(5), 257-269.
10. todman, l.c., bush, a., hood, a.s.c. (2023) 'small data' for big insights in ecology. trends ecol. evol. 38(7), 615-622.
11. youn, j., rai, n., tagkopoulos, i. (2022) knowledge integration and decision support for accelerated discovery of antibiotic resistance genes. nat commun. 13(1), 2360.
12. delios, a., clemente, e.g., wu, t., tan, h., wang, y., gordon, m., et al. (2022) examining the generalizability of research findings from archival data. proc. natl. acad. sci. u.s.a. 119(30), e2120377119.
13. chung, m.g., frank, k.a., pokhrel, y., dietz, t., liu, j. (2021) natural infrastructure in sustaining global urban freshwater ecosystem services. nat. sustain. 4, 1068-1075.
14. cooley, s.w. (2023) global loss of lake water storage. science 380(6646), 693.
15. cooley, s.w., ryan, j.c., smith, l.c. (2021) human alteration of global surface water storage variability. nature 591(7848), 78-81.
16. florke, m., schneider, c., mcdonald, r.i. (2018) water competition between cities and agriculture driven by climate change and urban growth. nat. sustain. 1(1), 51-58.
